
发布日期:2024-11-15 14:15:32
mRNA通过分子内碱基配对(二级结构)和高阶相互作用(三级结构)积极参与自我调节,形成复杂的结构。这些结构可以影响翻译的起始、延伸和终止,或作为调节细胞内翻译或mRNA衰变的蛋白质复合物的结合位点。未成对碱基的数量也是结构稳定性的衡量标准,用于指导序列设计。此外,在未配对的碱基中,未成对的U碱基最有可能影响序列的稳定性。因此,需要有效的方法优化mRNA结构,提高表达水平,并实现mRNA治疗药物的长期储存。
除了mRNA的序列结构优化外,最大限度减少体外转录(IVT)过程中产生的双链RNA(dsRNA)副产物同样对于mRNA治疗药物的稳定性和疗效至关重要。dsRNA副产物通过激活模式识别受体(PRR)来刺激宿主免疫反应,例如激活位于内体中的Toll样受体3(TLR3)、细胞质中的视黄酸诱导基因I(RIG-I)和黑色素瘤分化相关蛋白5(MDA-5)。此外,dsRNA还会激活RNA依赖性蛋白激酶R(PKR),导致翻译起始因子2(eIF-2α)的α亚基磷酸化,从而关闭蛋白合成。TLR3、PKR、OAS-RNase L和NLRP1等各种dsRNA受体的激活可导致诱导细胞死亡,由此激活的先天免疫反应不利于mRNA疗法的安全性和有效性。
2024年10月19日,连界启辰直投项目——艾博生物郜鹏团队联合同济大学生命科学与技术学院刘琦教授团队在期刊Journal of Molecular Biology上发表了题为“Engineered mRNAs with stable structures minimize double-stranded RNA formation and increase protein expression”的研究论文。这项研究揭示了序列中松散的mRNA二级结构和更多不成对的U碱基通常会导致在IVT过程中形成更多的dsRNA副产物。基于这些发现,进一步开发了一个基于序列特征的dsRNA副产物形成预测模型,以指导mRNA序列的优化,有助于最大限度地减少不需要的免疫反应并改善mRNA的蛋白质表达。这项研究为开发具有最少的dsRNA副产物和高蛋白质表达的mRNA疗法提供了新的线索和方法。

mRNA序列设计总结
本研究中选择了SARS-CoV-2变体BA.4 Spike RBD来研究mRNA序列与合成mRNA特征之间的关系,RBD蛋白包含241个氨基酸,对应于密码子组合约为10115。从该库中随机选择了100万个序列进行分析,以构建“随机”mRNA文库,检查密码子适应指数(CAI)、最小自由能(MFE)、尿嘧啶(U)数量和未配对的U数量等参数的分布,图1a显示了所选序列上述指标的特征聚类分布。
由于密码子使用频率(CUF)与宿主细胞中合成mRNA的表达水平密切相关,因此被认为是mRNA序列优化的关键因素。将从总mRNA库中选择的106个mRNA序列结合每个密码子的人类CUF值获得一个新的“CUF”库,与“随机”文库相比,这些序列在CAI、MFE、U数量和未配对的U数量方面显示出显著差异。
除了具有更高的CAI值外,与随机库相比,“CUF”库还表现出较低的MFE值,并且减少了总U和未配对的U数量。这一结果突出了CAI和MFE、总 U数量和未配对U数量之间的相关性,强调了将多个参数纳入序列优化分析的必要性。
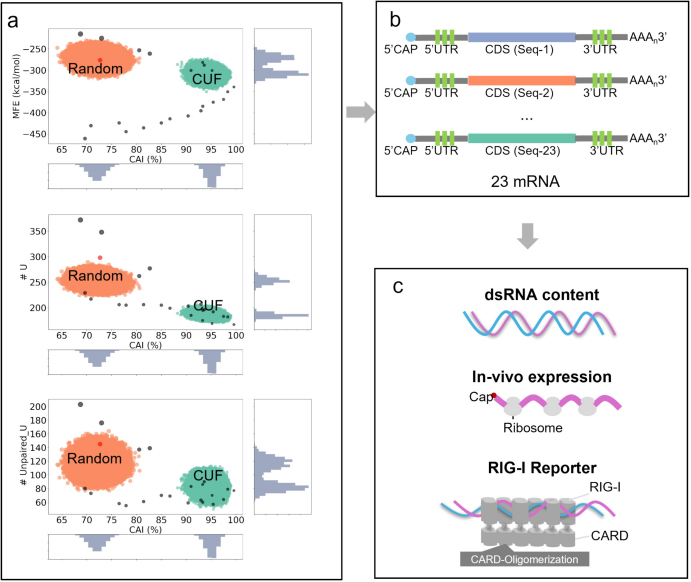
图1. 序列设计实验过程
基于上述结果,由于不同序列特征之间的相互关系,仅基于单个序列特征(例如CAI)的序列优化可能会限制RNA的整体设计空间,从而可能引入某些新的变量。为了解决这个问题,研究团队尝试引入多个变量来研究序列优化如何影响RNA产品质量。使用团队内部开发的密码子优化算法,首先设计了23个编码RBD的mRNA序列,在CAI、MFE、U数量和未配对的U数量上较前两个库表现出更广泛的多样性,涵盖了更广泛的设计范围。之后,将这些mRNA序列(图1b)合成并用于随后测定dsRNA含量、先天免疫反应(RIG-I)和体外表达水平(图1c)。
dsRNA水平与序列稳定性和U碱基比例有关
如图2a所示,23个mRNA的dsRNA含量差异高达20倍。进一步分析IVT mRNAs的dsRNA含量与其序列特性(CAI、MFE、U数量和未配对U数量)之间的关系。研究发现dsRNA含量与MFE和未配对U数量有一定程度的相关性(图2d、f)。相比之下,与总U数量的相关性较弱,与CAI值没有相关性(图2e、g)。因此,表明序列中松散的mRNA二级结构和更多不配对的U碱基导致在IVT过程中形成更多的dsRNA副产物(图2h)。
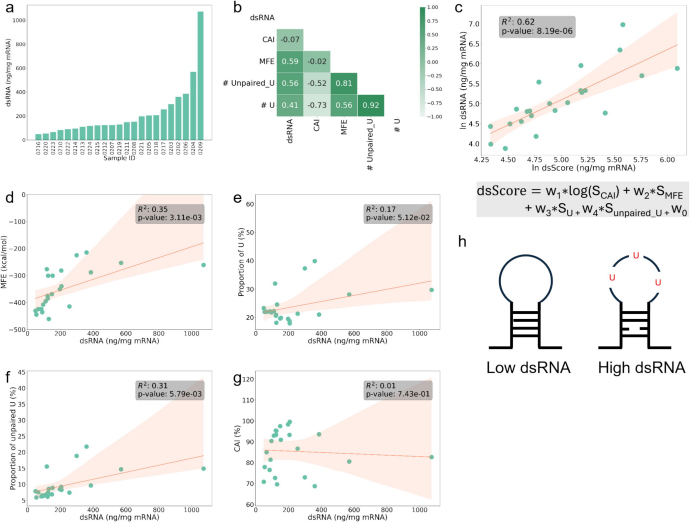
图2. dsRNA 含量与各序列特征之间的相关性
基于上述结果,研究团队进一步开发了一种基于mRNA序列预测IVT反应中dsRNA含量的算法——dsScore。该模型主要参考因素是未配对U的比例,dsScore与实际dsRNA含量呈明显的正相关(图2c),表明在IVT反应过程中产生dsRNA的潜在风险随着未配对U数量的减少而降低。
T7 RNA聚合酶与富含U的RNA结合更强
T7 RNA聚合酶通常用于通过体外转录(IVT)反应制备合成的mRNA。由于酶的性质,dsRNA副产物可以在反应过程中通过各种机制产生,例如RNA依赖性RNA复制的3'-延伸、转录本的不完全终止以及互补或回文序列的自退火。由于不良的启动子非依赖性转录可能始于EC(伸长复合体)构象,团队研究了T7 RNA聚合酶在EC构象中的结合状态。
使用含有不同比例U碱基的RNA:1U(5'-gggaaucgacagcgccgc-3',5.56%U)、5U(5'-gggaaucuaucuccgc-3',27.78% U)和9U(5'-gggaauuuuucucugc-3',50%U),发现RNA中U碱基的比例越高,与T7 RNA聚合酶的结合亲和力越强,这可能是促进RNA依赖性RNA聚合并促进dsRNA形成的原因(图3b)。分子动力学模拟确定5U和9U RNA结构与T7 RNA聚合酶复合物的ΔG值分别为-65 kcal/mol和-85 kcal/mol。对两种配合物表面相互作用的分析表明,虽然氢键的差异很小,但两种配合物内的离子键数量存在显著差异(图3a)。9U RNA与T7 RNAP形成6个离子键,而5U RNA形成3个离子键。这些离子键可能是影响RNA和T7 RNA聚合酶结合强度的关键因素。
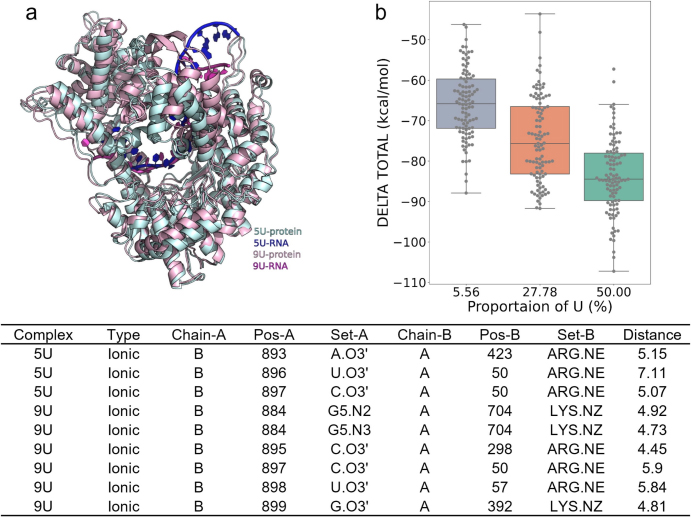
图3. U碱基数量对T7 RNA聚合酶结合亲和力的影响
dsRNA含量与RIG-I活化水平和体外表达相关
dsRNA杂质激活先天免疫系统可能导致外源性mRNA翻译的减少。前阶段研究中合成的编码RBD的23个mRNA表现出激活先天免疫的不同能力。研究发现dsRNA含量与RBD mRNA的RIG-I激活之间存在很强的相关性(皮尔逊相关系数R2= 0.58)(图4d)。其中,7个序列的蛋白表达水平低于野生型mRNA,而其余15个序列表现出更高的蛋白表达水平。最高和最低的蛋白质表达水平显示出近五倍的差异(图4b)。此外,RIG-I激活与蛋白质表达水平之间存在负相关(图4e)。综上所述,这些发现表明序列优化可能会减少IVT期间的dsRNA副产物,从而降低先天免疫反应,提高mRNA翻译水平(图4f)。
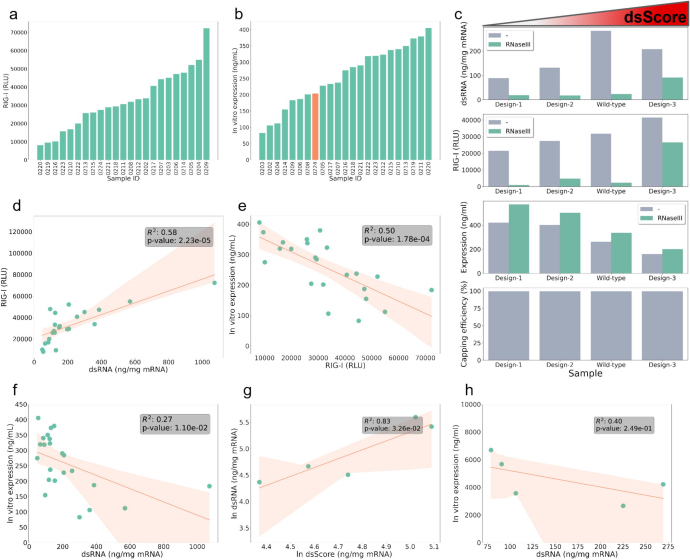
图4. 不同mRNA的RIG-I激活
为了验证dsScore算法的准确性并探索其在mRNA序列设计中降低dsRNA含量和提高蛋白质表达水平的应用,研究团队设计合成了四个dsRNA预测含量不同的mRNA序列(Design-1、Design-2、Design-3、野生型)。用RNase III处理后,dsRNA水平显著降低,RIG-I活化明显降低,伴随着蛋白质表达水平的增加(图4c)。令人惊讶的是,即使去除dsRNA后,四个样品的表达水平仍然不同,并且与dsScore呈负相关(图4c)。因此,序列特征会导致mRNA表达的差异并影响dsRNA含量;dsRNA的差异反过来会影响先天免疫激活,进而在一定程度上影响表达水平。
总结
本研究的重点是通过使用一致的IVT过程优化mRNA序列来调节dsRNA含量。尽管研究使用密码子优化mRNA验证了少数肽序列(Omicron BA.4变体,EPO)的模型,但这种方法也可能适用于不同的工艺条件,并适用于优化更广泛的蛋白质mRNA序列:
(1)具有更稳定结构的mRNA序列表现出较低的dsRNA含量和RIG-I激活减少,这与较高的体外蛋白表达水平相关;
(2) 序列中较高比例的未配对尿嘧啶(U)碱基导致dsRNA含量增加,从而确定有助于dsRNA生成的序列因子;
(3)IVT mRNA中dsRNA的含量可以通过基于mRNA结构信息的算法进行预测,为有效的mRNA序列设计提供新的指导。通过将该算法与密码子优化相结合,可以设计更稳定的RNA序列来控制dsRNA含量,从而减轻下游RNA纯化过程的负担。
本研究仅考虑了CAI、MFE、序列中U的比例以及未配对U的比例,这些序列特征与mRNA序列的稳定性和免疫原性密切相关,且是相互关联的。其他序列特征也可能与dsRNA含量有关,随着更多数据的引入,模型参数可能会发生变化。因此,持续迭代的mRNA序列优化技术可有效降低dsRNA含量,缓解下游纯化需求,同时减弱先天免疫反应,从而优化mRNA的生产和药物疗效。
参考资料
Qin Q, Yan H, Gao W, Cao R, Liu G, Zhang X, Wang N, Zuo W, Yuan L, Gao P, Liu Q. Engineered mRNAs with stable structures minimize double-stranded RNA formation and increase protein expression. J Mol Biol. 2024 Oct 18:168822. doi: 10.1016/j.jmb.2024.168822.

扫一扫关注公众号